สารบัญ:
- ผู้เขียน John Day day@howwhatproduce.com.
- Public 2024-01-30 13:03.
- แก้ไขล่าสุด 2025-01-23 15:12.
โดย: Phuc Lam, Paul Yeung, Eric Reyes
การยอมรับว่าข้อผิดพลาดในการแบ่งส่วนปอดจะทำให้เกิดข้อมูลเท็จเกี่ยวกับการระบุพื้นที่โรคและอาจส่งผลโดยตรงต่อกระบวนการวินิจฉัย เทคนิคการช่วยเหลือด้วยคอมพิวเตอร์สมัยใหม่ล้มเหลวในการให้ผลลัพธ์ที่แม่นยำเมื่อโรคปอดมีรูปร่างที่ท้าทาย รูปร่างที่ผิดปกติเหล่านี้อาจเกิดจากเยื่อหุ้มปอดไหลออก การรวมตัว ฯลฯ การใช้เทคนิคการแบ่งส่วนปอดซึ่งขอบเขตของปอดถูกแยกออกจากเนื้อเยื่อทรวงอกโดยรอบ แอปของเราสามารถระบุขอบเขตด้วยเกณฑ์การป้อนข้อมูลของผู้ใช้เพื่อให้มุมมองที่ปรับแต่งได้อย่างเต็มที่ ของรูปทรงของปอด
วัตถุประสงค์ของโครงการ MatLab นี้คือการสร้างแอปการแบ่งส่วนปอดแบบโต้ตอบที่ใช้งานง่าย เพื่อตรวจหาสภาวะทางพยาธิวิทยาของภาพเอ็กซ์เรย์ของปอด เป้าหมายของเราคือการสร้างวิธีที่มีประสิทธิภาพมากขึ้นในการแสดงตัวอย่างและระบุปอดที่ผิดปกติ เพื่อให้แพทย์และนักรังสีวิทยามีวิธีการวินิจฉัยโรคปอดที่เชื่อถือได้มากขึ้น การใช้เครื่องมือออกแบบแอปใน MatLab โปรแกรมได้รับการออกแบบมาเพื่อทำงานเฉพาะกับการสแกนเอ็กซ์เรย์ทรวงอกและการตรวจเอกซเรย์คอมพิวเตอร์ (CT) แต่ยังได้รับการทดสอบเพื่อทำงานกับการสแกนด้วย MRI
คำแนะนำด้านล่างประกอบด้วยเทคนิคการกรองสัญญาณรบกวนของเรา (ฟิลเตอร์ Wiener ความถี่ต่ำ) รวมถึงเกณฑ์ของภาพ (โดยใช้ฮิสโตแกรมความเข้มของภาพระดับสีเทา) และการใช้การไล่ระดับสีตามสัณฐานวิทยา (ความแตกต่างระหว่างการขยายและการพังทลายของภาพ) ถึง ระบุภูมิภาคที่น่าสนใจ คำแนะนำจะอธิบายวิธีที่เรารวมองค์ประกอบทั้งหมดเข้ากับอินเทอร์เฟซผู้ใช้แบบกราฟิก (GUI)
บันทึก:
1). โครงการนี้ได้รับแรงบันดาลใจจากงานวิจัยเรื่อง "การแบ่งส่วนและการวิเคราะห์ภาพของปอดผิดปกติที่ CT: แนวทางในปัจจุบัน ความท้าทาย และแนวโน้มในอนาคต" ซึ่งสามารถพบได้ที่นี่
2). เรากำลังใช้ภาพเอ็กซ์เรย์จาก NIH: Clinical Center ลิงค์ดูได้ที่นี่
3). ความช่วยเหลือเกี่ยวกับตัวออกแบบแอปสามารถพบได้ที่นี่
4). ก่อนรันโค้ด: คุณต้องเปลี่ยนเส้นทาง Dir (ในบรรทัดที่ 34) เป็นไดเร็กทอรีไฟล์และประเภทของภาพ (บรรทัดที่ 35) (เรากำลังวิเคราะห์ *.png)
ขั้นตอนที่ 1: ขั้นตอนที่ 1: กำลังโหลดรูปภาพ
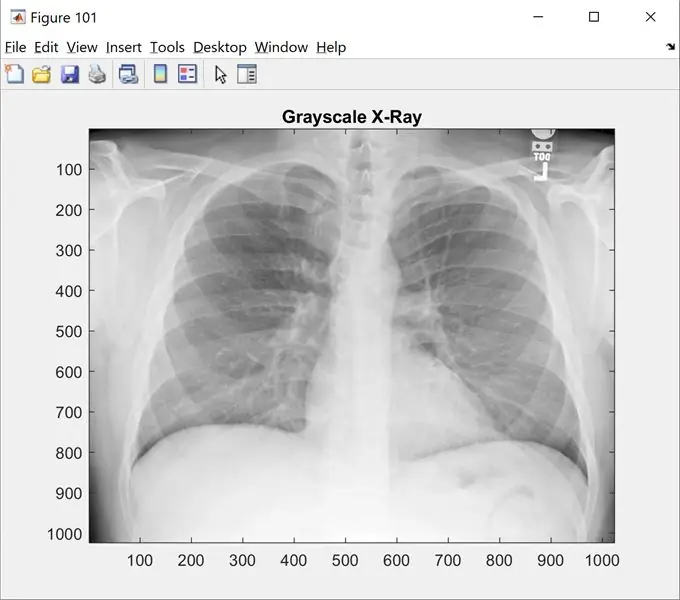
ขั้นตอนนี้จะแสดงรูปภาพต้นฉบับเป็นระดับสีเทา เปลี่ยน 'name_of_picture.png' เป็นชื่อรูปภาพของคุณ
แจ่มใส; ซีแอลซี; ปิดทั้งหมด;
%% กำลังโหลดรูปภาพ
raw_x_ray='name_of_picture.png';
ฉัน=imread(raw_x_ray);
รูป(101);
อิมโชว์(I);
แผนที่สี(สีเทา);
title('เอกซเรย์สีเทา');
ขั้นตอนที่ 2: ขั้นตอนที่ 2: การกรองสัญญาณรบกวนและฮิสโตแกรม
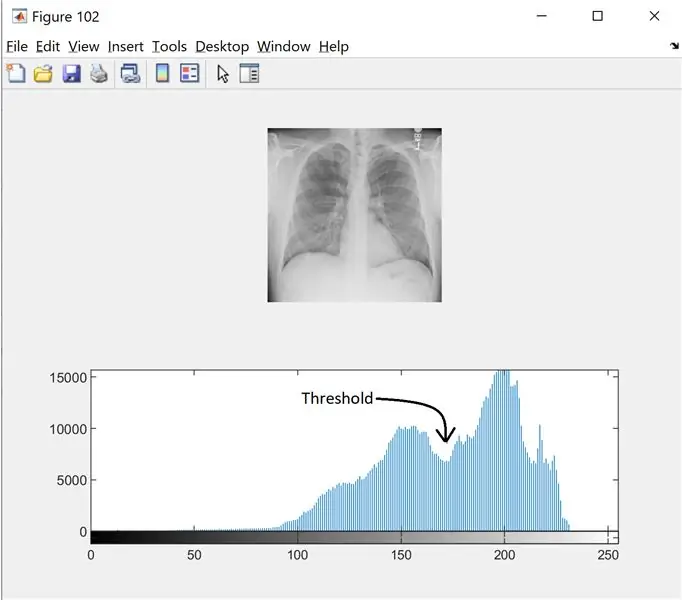
ในการหาขีดจำกัดของภาพระดับสีเทา เราจะดูที่ฮิสโตแกรมเพื่อดูว่ามีโหมดที่แตกต่างกันหรือไม่ อ่านเพิ่มเติมได้ที่นี่
ผม=wiener2(ผม, [5 5]);
รูป(102);
แผนย่อย(2, 1, 1);
อิมโชว์(I);
แผนย่อย(2, 1, 2);
imhist(I, 256);
ขั้นตอนที่ 3: ขั้นตอนที่ 3: การตั้งค่าเกณฑ์
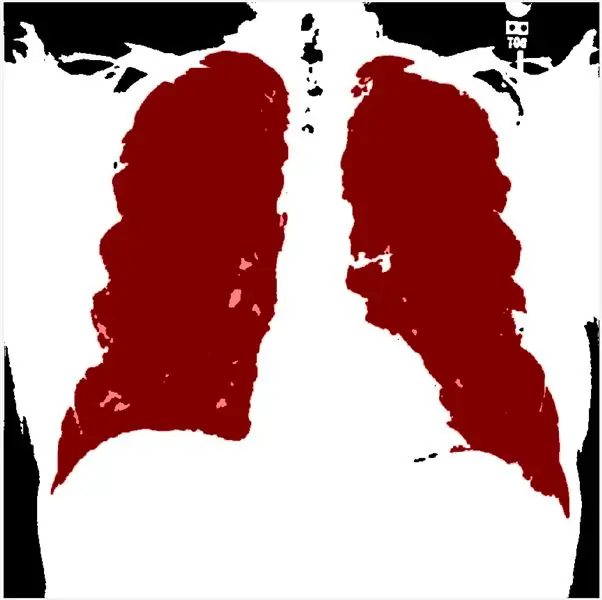
ขั้นตอนนี้ช่วยให้คุณกำหนดเกณฑ์ตามฮิสโตแกรมได้ morphologicalGradient จะเน้นบริเวณที่สนใจเป็นสีแดง และฟังก์ชัน visboundaries จะซ้อนทับภาพที่ร่างและกรองของปอดเป็นสีแดง
ด้วยการใช้ regionprops เราสามารถกำหนดอาร์เรย์ของความแข็งแกร่งและจัดเรียงจากมากไปน้อยได้ ต่อไป ฉันสร้างไบนารีภาพสเกลสีเทาและใช้วิธีไล่ระดับสัณฐานวิทยาและ mLoren Shurasking เพื่อเน้นพื้นที่ที่น่าสนใจ (ROI) ขั้นตอนต่อไปคือการกลับภาพเพื่อให้ ROI ของปอดเป็นสีขาวบนพื้นหลังสีดำ ฉันใช้ฟังก์ชัน showMaskAsOverlay เพื่อแสดง 2 mask หมายเหตุ: รหัสนี้ได้รับแรงบันดาลใจจาก Loren Shure ลิงก์
สุดท้ายนี้ ฉันสร้างโครงร่างสีแดงโดยใช้ bwbwboundaries และปิดบังภาพตัวกรองและขอบเขต
a_thresh = ฉัน >= 172; % กำหนดเกณฑ์นี้
[labelImage, numberOfBlobs] = bwlabel(a_thresh);
props = regionprops (a_thresh, 'ทั้งหมด');
sortedSolidity = sort ([props. Solidity], 'ลง');
SB = sortedSolidity (1);
ถ้า SB == 1% SB รับแต่ความแน่น == 1 กรองกระดูก
binaryImage = imbinarize (ฉัน); รูป(103);
imshow (ภาพไบนารี); แผนที่สี(สีเทา);
SE = strel('สี่เหลี่ยม', 3);
morphologicalGradient = imsubtract(imdilate(binaryImage, SE), imerode(binaryImage, SE));
หน้ากาก = imbinarize(morphologicalGradient, 0.03);
SE = strel('สี่เหลี่ยม', 2);
หน้ากาก = ปิด (หน้ากาก SE);
หน้ากาก = imfill(หน้ากาก 'หลุม');
หน้ากาก = bwareafilt(หน้ากาก 2); % ควบคุมจำนวนพื้นที่แสดง
notMask = ~หน้ากาก;
หน้ากาก = หน้ากาก | bwpropfilt(notMask, 'พื้นที่', [-Inf, 5000 - eps(5000)]);
showMaskAsOverlay(0.5, หน้ากาก, 'r'); % คุณต้องดาวน์โหลดแอป/ฟังก์ชั่น showMaskAsOverlay
BW2 = imfill (ภาพไบนารี 'หลุม');
new_image = BW2;
new_image(~mask) = 0; % พลิกพื้นหลังและรู
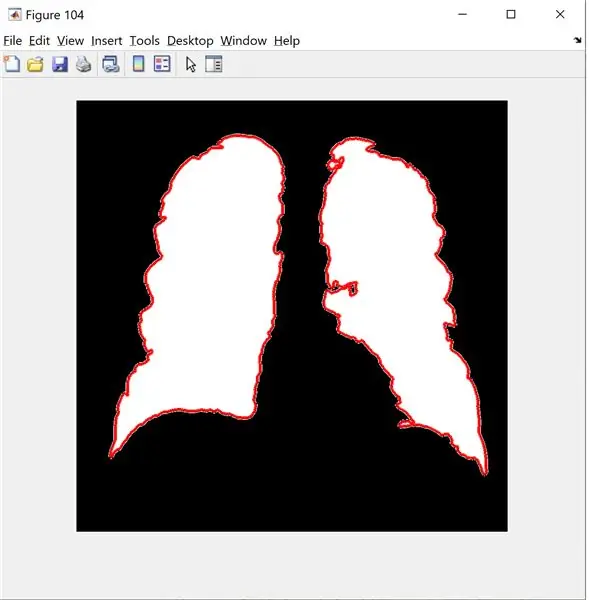
B=bwboundaries(new_image); % รับได้เพียง 2 มิติเท่านั้น
รูป(104);
imshow(new_image);
เดี๋ยว
visboundaries(B);
จบ
ขั้นตอนที่ 4: การสร้าง GUI
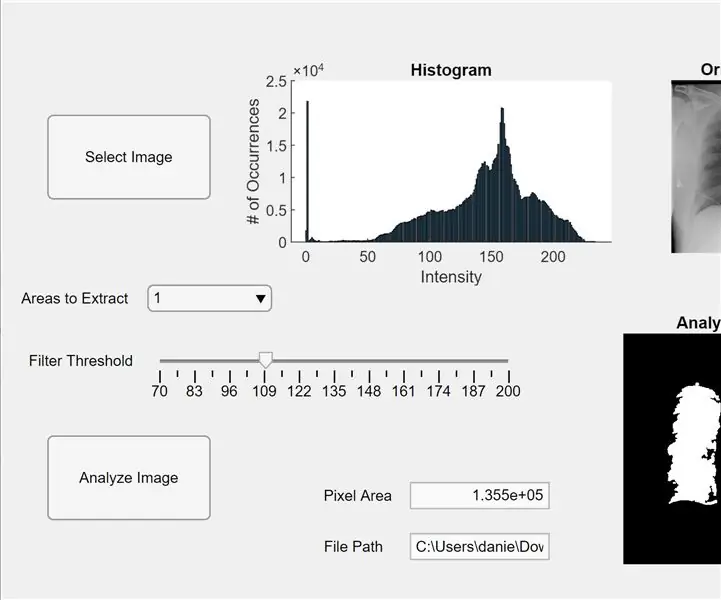
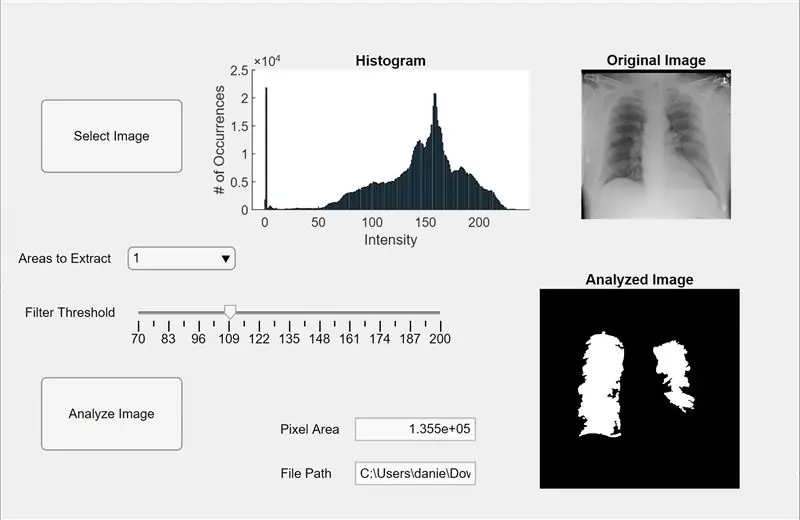
ตอนนี้ เรารวมโค้ดก่อนหน้าเข้ากับแอป MATLAB เปิดตัวออกแบบแอปใน MATLAB (ใหม่ > แอป) ขั้นแรก เราออกแบบอินเทอร์เฟซโดยคลิกค้างไว้แล้วลากสามแกนลงในพื้นที่ทำงานตรงกลาง ต่อไป เราคลิกค้างไว้แล้วลากสองปุ่ม หนึ่งฟิลด์แก้ไข (ข้อความ) หนึ่งฟิลด์แก้ไข (ตัวเลข) หนึ่งตัวเลื่อน และหนึ่งเมนูดรอปดาวน์ สองแกนจะแสดงภาพตัวอย่างและวิเคราะห์ภาพ และแกนที่สามจะแสดงฮิสโตแกรมของพิกเซลสำหรับภาพตัวอย่าง "ที่เลือก" ช่องแก้ไข (ข้อความ) จะแสดงเส้นทางไฟล์ของรูปภาพที่เลือก และช่องแก้ไข (ตัวเลข) จะแสดงพื้นที่พิกเซลที่ตรวจพบของปอด
ตอนนี้เปลี่ยนจากมุมมองการออกแบบเป็นมุมมองโค้ดใน App Designer ป้อนรหัสรหัสสำหรับคุณสมบัติโดยคลิกปุ่ม "คุณสมบัติ" สีแดงพร้อมเครื่องหมายบวก เริ่มต้นคุณสมบัติ I, threshold, และ routesToExtract ตามโค้ดด้านล่าง ถัดไป ให้คลิกขวาที่ปุ่มที่ด้านขวาบนของเวิร์กสเปซ (เบราว์เซอร์คอมโพเนนต์) และไปที่ Callbacks>Go to… callback เพิ่มรหัสสำหรับ “ฟังก์ชั่น SelectImageButtonPushed (แอพ, เหตุการณ์)” รหัสนี้ให้คุณเลือกรูปภาพเพื่อวิเคราะห์จากคอมพิวเตอร์ของคุณโดยใช้ uigetfile หลังจากเลือกรูปภาพแล้ว ภาพตัวอย่างจะปรากฏใต้แกนพร้อมกับฮิสโตแกรม จากนั้นให้คลิกขวาที่ปุ่มอื่นและทำซ้ำขั้นตอนเดิมเพื่อสร้างฟังก์ชันเรียกกลับ
เพิ่มโค้ดใต้ "ฟังก์ชัน AnalyzeImageButtonPushed (แอป เหตุการณ์)" โค้ดนี้จะดำเนินการนับพิกเซลและตรวจจับหยดบนภาพตัวอย่างโดยใช้ปุ่มวิเคราะห์ภาพ (แล้วแต่ว่าคุณจะคลิกขวาที่โค้ดใดสำหรับโค้ดนี้) หลังจากตั้งโปรแกรมปุ่มแล้ว เราจะตั้งโปรแกรมตัวเลื่อนและเมนูแบบเลื่อนลง คลิกขวาที่ตัวเลื่อน สร้างฟังก์ชันเรียกกลับ และเพิ่มในโค้ดใต้ "ฟังก์ชัน FilterThresholdSliderValueChanged (แอป เหตุการณ์)" จนจบ ซึ่งช่วยให้แถบเลื่อนปรับเกณฑ์ความเข้มของสีเทาได้
สร้างฟังก์ชันเรียกกลับสำหรับเมนูแบบเลื่อนลง และเพิ่มโค้ดใต้ "ฟังก์ชัน AreastoExtractDropDownValueChanged (แอป เหตุการณ์)" เพื่อให้เมนูดรอปดาวน์แก้ไขจำนวน blobs ที่แสดงบนแกนภาพที่วิเคราะห์ได้ ตอนนี้ คลิกแต่ละเอนทิตีในเบราว์เซอร์คอมโพเนนต์แล้วเปลี่ยนคุณสมบัติตามที่คุณต้องการ เช่น เปลี่ยนชื่อของเอนทิตี ลบแกน และเปลี่ยนมาตราส่วน ลากและวางเอนทิตีของเบราว์เซอร์คอมโพเนนต์ในมุมมองออกแบบไปยังเลย์เอาต์ที่ใช้งานได้จริงและเข้าใจง่าย ตอนนี้คุณมีแอพใน MATLAB ที่สามารถวิเคราะห์ภาพของปอดสำหรับพื้นที่พิกเซลได้แล้ว!
คุณสมบัติ (การเข้าถึง = ส่วนตัว)ฉัน = ; % ไฟล์ภาพ
เกณฑ์ = 257; %threshold สำหรับการแบ่งระดับความเข้มของสีเทา
RegionToExtract = 2;
จบ
ฟังก์ชั่น SelectImageButtonPushed (แอพ, เหตุการณ์)
clc;Dir = 'C:\Users\danie\Downloads\images_004\images'; %define ไฟล์ invariate "คำนำหน้า"
[imageExt, เส้นทาง] = uigetfile('*.png'); % คว้าส่วนตัวแปรของชื่อภาพ
imageName = [ไฟล์ Direp imageExt]; % concatenate invariate และสายพันธุ์ที่แปรผันได้
app. I = imread (ชื่อรูปภาพ); %อ่านภาพ
imshow(app. I, 'parent', app. UIAxes); %แสดงภาพ
app. FilePathEditField. Value = เส้นทาง; % แสดงเส้นทางไฟล์ของที่มาของภาพต้นฉบับ
จบ
ฟังก์ชัน AnalyzeImageButtonPushed (แอป เหตุการณ์)
ภาพต้นฉบับ = app. I;
ภาพต้นฉบับ = wiener2(app. I, [5 5]); %ตัวกรองการลบจุด
ฮิสโตแกรม (app. AxesHistogram, app. I, 256); %แสดงฮิสโตแกรมของภาพ
a_thresh = originalImage >= app.threshold; % กำหนดเกณฑ์นี้
labelImage = bwlabel(a_thresh);
props = regionprops (a_thresh, 'ทั้งหมด');
sortedSolidity = sort ([props. Solidity], 'ลง');
SB = sortedSolidity (1);
ถ้า SB == 1% SB ยอมรับเฉพาะความแข็งแกร่ง ==1 กรองกระดูกออก
SE = strel('สี่เหลี่ยม', 3);
morphologicalGradient = imsubtract(imdilate(labelImage, SE), imerode(labelImage, SE));
หน้ากาก = imbinarize(morphologicalGradient, 0.03);
SE = strel('สี่เหลี่ยม', 2);
หน้ากาก = ปิด (หน้ากาก SE);
หน้ากาก = imfill(หน้ากาก 'หลุม');
mask = bwareafilt (หน้ากาก, app.regionsToExtract);
% ควบคุมจำนวนพื้นที่แสดง
notMask = ~หน้ากาก;
หน้ากาก = หน้ากาก | bwpropfilt(notMask, 'พื้นที่', [-Inf, 5000 - eps(5000)]);
BW2 = imfill (labelImage, 'holes');
new_image = BW2;
new_image(~mask) = 0;
B = bwboundaries(new_image); % ยอมรับได้เพียง 2 มิติเท่านั้น imshow (new_image, 'parent', app. UIAxes2);
ถือ (app. UIAxes2, 'เปิด');
visboundaries(B);
set(gca, 'YDir', 'reverse');
lungArea = bwarea(new_image);
app. PixelAreaEditField. Value = lungArea; แอป
จบ
จบ
ฟังก์ชัน FilterThresholdSliderValueChanged (แอป เหตุการณ์)
app.threshold = app. FilterThresholdSlider. Value;
จบ
ฟังก์ชัน AreastoExtractDropDownValueChanged (แอป เหตุการณ์) stringNumber = app. AreastoExtractDropDown. Value;
app.regionsToExtract = str2double(stringNumber);
จบ
จบ
แนะนำ:
MATLAB การตรวจจับใบหน้าอย่างง่าย: 4 ขั้นตอน

MATLAB Easy Face Detection: จุดประสงค์หลักของคำแนะนำนี้คือเพื่อแสดงให้เห็นว่าการประมวลผลภาพจะง่ายเพียงใด ด้วยความช่วยเหลือของ MATLABFace การตรวจจับและติดตามเป็นสาขาการวิจัยที่สำคัญและกระตือรือร้น นั่นคือเหตุผลที่ฉันจะอธิบาย จะทำได้อย่างไร
วิธีเขียนโค้ดและเผยแพร่ Matlab 2016b เป็น Word (คู่มือสำหรับผู้เริ่มต้น): 4 ขั้นตอน

How to Code and Publish Matlab 2016b to Word (Beginners Guide): Matlab เป็นโปรแกรมภาษาประสิทธิภาพสูงที่ใช้ในการคำนวณผลลัพธ์ทางเทคนิค มีความสามารถในการรวมภาพ การคำนวณ และการเขียนโปรแกรมในลักษณะที่เป็นมิตรต่อผู้ใช้ ด้วยโปรแกรมนี้ ผู้ใช้สามารถเผยแพร่ปัญหาและแนวทางแก้ไข
การควบคุม Raspberry Pi โดยใช้ Matlab: 5 ขั้นตอน

การควบคุม Raspberry Pi โดยใช้ Matlab: เฮ้ บทช่วยสอนนี้เกี่ยวกับการควบคุม Raspberry Pi ที่ไม่มีหัวของคุณโดยใช้ Matlab คุณอาจต้องติดตั้ง matlab เป็นเวอร์ชันล่าสุดเพื่อรองรับบอร์ด raspberry pi ที่ใหม่กว่า
ROS Robotic Controller ที่ใช้ Matlab: 9 ขั้นตอน
ROS Robotic Controller ที่ใช้ Matlab: ตั้งแต่ฉันยังเป็นเด็ก ฉันใฝ่ฝันที่จะเป็น Iron Man และยังคงทำเช่นนั้น Iron Man เป็นหนึ่งในตัวละครเหล่านั้นซึ่งเป็นไปได้อย่างสมจริง และเพียงแค่ทำให้ฉันปรารถนาที่จะเป็น Iron Man สักวันหนึ่ง แม้ว่าผู้คนจะหัวเราะเยาะฉันหรือบอกว่ามันเป็นไปไม่ได้
ฝึกการเพิ่มประสิทธิภาพการเขียนโปรแกรมใน MATLAB: 6 ขั้นตอน

Train Programming Enhancements ใน MATLAB: วัตถุประสงค์:เป้าหมายของระบบที่ตั้งโปรแกรมไว้นี้คือการพิจารณา Arduino ขนาดเล็ก และใช้การเข้ารหัสในขนาดที่ใหญ่ขึ้น เพื่อเพิ่มศักยภาพด้านความปลอดภัยบางอย่างของระบบ Amtrak Railroad ในการทำเช่นนี้ เราได้เพิ่มความชื้นในดิน sen
