
สารบัญ:
- ขั้นตอนที่ 1: ค้นหาการสแกนด้วย MRI และ MRA ของสมองปกติและสมองที่ได้รับผลกระทบจาก Moyamoya
- ขั้นตอนที่ 2: โหลดรูปภาพลงใน MATLAB และกำหนดรูปภาพให้กับตัวแปรเพื่อแสดงรูปภาพ
- ขั้นตอนที่ 3: ปรับปรุงโครงสร้างที่ยืดออกในรูปภาพที่มีความเข้มด้วยการกรองแบบหลายสเกล
- ขั้นตอนที่ 4: เรียกใช้ตัวกรองค่ามัธยฐาน 2 มิติ
- ขั้นตอนที่ 5: ปิดบังภาพ
- ขั้นตอนที่ 6: เลือก MRA Scans สำหรับการทดสอบทางสถิติ
- ขั้นตอนที่ 7: คำนวณพื้นที่ของหลอดเลือดในการเตรียมการทดสอบทางสถิติ
- ขั้นตอนที่ 8: เรียกใช้ตัวอย่างอิสระ T-test
- ผู้เขียน John Day day@howwhatproduce.com.
- Public 2024-01-30 13:07.
- แก้ไขล่าสุด 2025-01-23 15:12.

Moyamoya "พัฟควัน" เป็นโรคหายากที่เกิดจากการอุดตันของหลอดเลือดแดงที่ปมประสาทฐานซึ่งเป็นพื้นที่ที่ฐานของสมอง โรคนี้เป็นโรคหลอดเลือดสมองที่มีความก้าวหน้าซึ่งส่วนใหญ่ส่งผลกระทบต่อเด็ก อาการของโมยาโมยา ได้แก่ จังหวะเริ่มต้น จังหวะสั้นๆ อย่างต่อเนื่อง กล้ามเนื้ออ่อนแรง อัมพาต หรืออาการชักอันเป็นผลมาจากการตีบตันของหลอดเลือดแดง หากไม่ได้รับการรักษา โมยาโมยาจะทำให้เกิดปัญหาในการพูด ความบกพร่องทางประสาทสัมผัส และสติสัมปชัญญะ ในโครงการของเรา เราจะใช้ MATLAB เพื่อประมวลผลภาพ MRI หรือ MRA ล่วงหน้าโดยใช้ตัวกรองต่างๆ เพื่อลดสัญญาณรบกวนภายในภาพเพื่อค้นหาพื้นที่ที่ได้รับผลกระทบ นอกจากนี้ เราจะใช้การปรับปรุงคุณสมบัติเพื่อค้นหาพื้นที่ที่ได้รับผลกระทบได้แม่นยำยิ่งขึ้น ยิ่งไปกว่านั้น เราจะทำการทดสอบตัวอย่างอิสระเพื่อดูว่ามีความแตกต่างอย่างมีนัยสำคัญระหว่างปริมาณของหลอดเลือดในสมองปกติหรือไม่เมื่อเทียบกับสมองที่ได้รับผลกระทบจากโมยาโมยา
ขั้นตอนที่ 1: ค้นหาการสแกนด้วย MRI และ MRA ของสมองปกติและสมองที่ได้รับผลกระทบจาก Moyamoya




ภาพเหล่านี้เป็นภาพสแกนที่เราใช้สำหรับโครงการที่เราพบทางออนไลน์ ภาพสองภาพที่มีหลอดเลือดอยู่ตรงกลางคือการสแกน MRA ในขณะที่อีกสองภาพคือการสแกนด้วย MRI
ลิงค์ต่อไปนี้คือตำแหน่งที่พบภาพเหล่านี้:
static.cambridge.org/resource/id/urn:cambr…
static.cambridge.org/resource/id/urn:cambr…
ขั้นตอนที่ 2: โหลดรูปภาพลงใน MATLAB และกำหนดรูปภาพให้กับตัวแปรเพื่อแสดงรูปภาพ

ในการเริ่มต้นกระบวนการ ให้เริ่มต้นด้วยการล้างหน้าต่างคำสั่ง ปิดตัวเลขและกราฟที่เป็นไปได้ทั้งหมดที่อาจเปิดอยู่แล้วและล้างตัวแปรที่กำหนดไว้แล้วในพื้นที่ทำงานของคุณ
หลังจากนั้น สร้าง for loop จาก 1 ถึง 2 โดยใช้คำสั่ง i = [1:2]
หลังจากนั้น โหลดอิมเมจ MRA โดยใช้คำสั่ง imread(sprintf('filename%.filetype', i)) เพื่ออ่านรูปภาพจากไฟล์ที่ระบุโดยชื่อไฟล์ ตามด้วยหมายเลขที่ใช้สำหรับลูปโดยใช้ sprintf สำหรับการโหลดและกำหนดแบตช์ ให้เป็นตัวแปร
จากนั้นเพื่อแสดงรูปภาพในรูป ให้ใช้คำสั่ง imshow(I)
ในการกำหนด colormap สีเทา ให้ใช้คำสั่ง colormap(gray)
ในการกำจัดสีและแปลงเมทริกซ์ 3 มิติสำหรับรูปภาพเป็น 2 มิติอย่างสมบูรณ์ ให้ใช้คำสั่ง rgb2gray(I) และกำหนดให้กับตัวแปรแยกต่างหาก
จากนั้นโหลดอิมเมจ MRI โดยใช้คำสั่งที่ระบุก่อนหน้านี้หรือ imread(sprintf('filename%.filetype', i)) และกำหนดให้กับตัวแปรใหม่
ทำซ้ำคำสั่ง rgb2gray ด้วยตัวแปรใหม่ที่ใช้สำหรับอิมเมจ MRI
หากจำเป็น คุณสามารถปรับขนาดรูปภาพโดยใช้คำสั่ง imresize(A, scale) และกำหนดให้กับตัวแปรแยกต่างหาก
ขั้นตอนที่ 3: ปรับปรุงโครงสร้างที่ยืดออกในรูปภาพที่มีความเข้มด้วยการกรองแบบหลายสเกล

การใช้ตัวแปรใหม่ ใช้คำสั่ง fibermetric(A) เพื่อปรับปรุงโครงสร้างท่อในภาพ
เมื่อใช้ตัวแปรก่อนหน้านี้ ให้ใช้คำสั่ง histeq(B) เพื่อปรับปรุงการปรับสมดุลฮิสโตแกรมโดยเปลี่ยนความเข้มของภาพและกำหนดให้กับตัวแปรใหม่
แสดงฮิสโตแกรมโดยใช้คำสั่ง imhist(B)
สร้างตัวแปรใหม่เพื่อสร้างขีดจำกัดสำหรับตัวกรอง ในกรณีนี้กำหนดตัวแปรก่อนหน้า> 0.875 โดยกรองความเข้มของพิกเซลออกภายใต้ค่า 0.875
หลังจากนั้น ให้สร้างฟิกเกอร์ใหม่และใช้คำสั่ง imshow(A) เพื่อแสดงภาพที่กรองแล้วใหม่
ขั้นตอนที่ 4: เรียกใช้ตัวกรองค่ามัธยฐาน 2 มิติ

การใช้คำสั่ง medfilt2(A, [mn]) ให้รันตัวกรองค่ามัธยฐาน 2D โดยที่แต่ละพิกเซลเอาต์พุตมีค่ามัธยฐานในขอบเขต mxn รอบพิกเซลที่เกี่ยวข้องในภาพอินพุต
สร้างตัวเลขใหม่และใช้ imshow(A) เพื่อแสดงภาพที่กรองค่ามัธยฐาน
ขั้นตอนที่ 5: ปิดบังภาพ

การใช้ภาพกรองกลาง ใช้คำสั่ง [labeledImage, numberOfBlots] = bwlabel(A) เพื่อนับจำนวนจุดสีขาวในภาพ
จากนั้นใช้ฟังก์ชัน region props state = regionprops (labeledImage, 'Area') เพื่อคำนวณพื้นที่ของแต่ละ blot หรือหลอดเลือด
กำหนดพื้นที่ทั้งหมดให้เป็นตัวแปรเดียว
จากนั้นใช้ตัวแปรอื่น นับจำนวนจุดบล๊อกที่เกิน 50 พิกเซล
หลังจากนั้น ให้จัดเรียงจุดที่มีจุดต่ำกว่า 50 พิกเซลโดยเรียงลำดับจากมากไปน้อยโดยใช้คำสั่ง [sortedAreas, sortedIndicies] = sort(Areas, 'descend')
จากนั้นใช้ตัวแปรอื่นใช้คำสั่ง ismember(labeledImage, sortedIndicies(1:numberToExtract)) เพื่อส่งคืนอาร์เรย์ที่มีองค์ประกอบของ labeledImage อยู่ใน sortedIndicies จากหมายเลข 1 ถึงจำนวนหลอดเลือดเพื่อส่งคืนตรรกะ 1 (จริง) หรือ ตรรกะ 0 (เท็จ)
ด้วยตัวแปรในขั้นตอนก่อนหน้า ให้ค้นหาจุดที่เป็นจริง (ค่า > 0) และสร้างอาร์เรย์แบบลอจิคัลเพื่อสร้างอิมเมจไบนารีและกำหนดให้กับตัวแปรใหม่
สร้างตัวเลขใหม่และใช้ imshow(A) อิมเมจไบนารีใหม่
จากนั้นให้กลับภาพโดยใช้คำสั่ง imcomplement(A) และกำหนดให้กับตัวแปรอื่น
ในการสร้างภาพมาสก์ ให้ใช้ตัวแปรใหม่พร้อมคำสั่ง resizedimage.*uint8(invertedimage)
สร้างร่างใหม่และใช้ imshow(A) เพื่อแสดงภาพที่ปิดบัง
หากต้องการจบโค้ดทั้งหมด ให้ใช้คำสั่ง 'end' เพื่อสิ้นสุดโค้ดทั้งหมดสำหรับลูป
ขั้นตอนที่ 6: เลือก MRA Scans สำหรับการทดสอบทางสถิติ



ในการเตรียมตัวสำหรับการทดสอบทางสถิติ ให้เลือกการสแกน MRA ที่จะใช้สำหรับการทดสอบตัวอย่างอิสระ เนื่องจากสองตัวอย่างของเราจะเป็นสมองที่ได้รับผลกระทบ Moyamoya และสมองปกติ ให้เลือกการสแกน MRA ในแต่ละกลุ่มในปริมาณที่เหมาะสม
ขั้นตอนที่ 7: คำนวณพื้นที่ของหลอดเลือดในการเตรียมการทดสอบทางสถิติ

การทดสอบทางสถิติจะเน้นที่ความยาวหรือปริมาณของหลอดเลือดที่แสดงในการสแกน MRA ดังนั้นเราต้องคำนวณพื้นที่ของหลอดเลือดก่อนเปรียบเทียบ
เริ่มต้นด้วยการกรอง MRA ของสมองปกติและคำนวณปริมาณของหลอดเลือด เมื่อต้องการทำสิ่งนี้ ให้รัน for loop เนื่องจากมีสามภาพ เงื่อนไขจะเป็น i = [1:3]
เปิดอิมเมจด้วยคำสั่ง imread และกำหนดให้กับตัวแปร
จากนั้น สร้างคำสั่ง if/else ด้วยคำสั่ง if, else สำหรับคำสั่ง if ให้ใช้คำสั่ง size(A, 3)==3 โดยที่ A เป็นตัวแปรที่ใช้เปิดรูปภาพ เพื่อสร้างคำสั่ง if เมื่อมิติที่ 3 ของอาร์เรย์เป็น 3 แล้วแปลงอิมเมจ เป็น 2D และกำจัดสีโดยใช้คำสั่ง rgb2gray(A) และกำหนดให้กับตัวแปรใหม่ ใช้คำสั่ง imresize(A, [mn]) เพื่อปรับขนาดภาพ ในกรณีนี้ เราปรับขนาดรูปภาพเป็น 1024 x 1024 เมทริกซ์ ในการปรับปรุงโครงสร้างท่อของรูปภาพ ให้ใช้คำสั่ง fibermetric อีกครั้ง และกำหนดให้กับตัวแปรใหม่
ต่อไปนี้สำหรับคำสั่ง else หากรูปภาพไม่ใช่เมทริกซ์ 3 มิติ เราต้องการข้ามการแปลง ทำเช่นเดียวกับคำสั่ง if แต่ไม่มีคำสั่ง rgb2gray(A)
สร้างตัวแปรใหม่ โดยตั้งค่าให้เท่ากับตัวแปรจากขั้นตอนไฟเบอร์เมตริกที่มากกว่า 0.15 เกณฑ์นี้กำหนดอิมเมจสำหรับความเข้มที่มากกว่า 0.15
เราจะทำซ้ำบรรทัดของรหัสจากขั้นตอนที่ 4 และ 5 ของคำสั่งจากบรรทัดตัวกรองค่ามัธยฐานจนถึงบรรทัด imshow(I) หลังจากนั้น ใช้คำสั่ง sum(I(:)) เพื่อเพิ่มพิกเซลทั้งหมดที่ประกอบเป็นเส้นเลือด และกำหนดให้กับตัวแปรแยกต่างหาก ตั้งชื่อตัวแปรใหม่ NormalBloodVessels(i) และตั้งค่าให้เท่ากับตัวแปรจากคำสั่ง sum(I(:)) สิ่งนี้จะเพิ่มข้อมูลลงในเมทริกซ์
จบการวนซ้ำและทำซ้ำ แต่สำหรับ MRA ของสมองที่ได้รับผลกระทบจาก Moyamoya ตั้งชื่อตัวแปรในตอนท้าย MoyaMoyaBloodVessels(i) เพื่อไม่ให้สับสนกับ MRA ของสมองปกติ
ขั้นตอนที่ 8: เรียกใช้ตัวอย่างอิสระ T-test

เนื่องจากมีตัวอย่างอิสระสองตัวอย่าง และประชากรจำนวนน้อย ให้รันตัวอย่างอิสระ t-test
สร้างและตั้งชื่อฟังก์ชันที่ทำการทดสอบตัวอย่างอิสระเพื่อตรวจสอบว่าปริมาณหลอดเลือดใน MRA ของสมองปกติมีค่าเท่ากับ MRA ของสมองที่ได้รับผลกระทบจาก Moyamoya อย่างมีนัยสำคัญหรือไม่
แสดงชุดสมมติฐานสำหรับการทดสอบโดยใช้คำสั่ง disp('X') ในบรรทัดแรก ให้แสดง "สมมติฐานสำหรับการทดสอบตัวอย่างสองตัวอย่าง" ในบรรทัดที่สอง ให้แสดง "H0 = ปริมาณหลอดเลือดในสมองปกติเท่ากับปริมาณหลอดเลือดในสมองที่เป็นโรค Moyamoya" เพื่อระบุสมมติฐานว่าง ในบรรทัดที่สาม แสดง "HA = ปริมาณหลอดเลือดในสมองปกติไม่เท่ากับปริมาณหลอดเลือดในสมองที่เป็นโรค Moyamoya" เพื่อระบุสมมติฐานทางเลือก
ใช้ช่วงความเชื่อมั่น 95% และขนาดกลุ่มตัวอย่าง 3 คำนวณคะแนน t โดยใช้คำสั่ง tinv([0.025 0.975], 2) และกำหนดให้กับตัวแปร t ใช้คำสั่ง var(NormalBloodVessels) และ var(MoyaMoyaBloodVessels) และกำหนดให้กับตัวแปรเพื่อคำนวณความแปรปรวนของชุดข้อมูลทั้งสองชุด
ตรวจสอบว่าความแปรปรวนใกล้เคียงกันหรือไม่ ทำได้โดยการสร้างคำสั่ง if/else ด้วยคำสั่ง if, else สำหรับเงื่อนไขในคำสั่ง if ให้เขียน A / B == [0.25:4] โดยที่ A คือตัวแปรที่อธิบายความแปรปรวนของหลอดเลือดปกติ และ B คือตัวแปรที่อธิบายความแปรปรวนของหลอดเลือด Moyamoya 0.25 และ 4 มาจากค่าประมาณทั่วไปในการพิจารณาว่าความแปรปรวนเท่ากันหรือไม่ จากนั้นให้รันสองตัวอย่าง t test ด้วย [h, p] = ttest2(A, B, 0.05, 'both', 'equal') โดยที่ A และ B เป็นตัวแปรเดียวกันกับที่กล่าวไว้ก่อนหน้านี้ สำหรับคำสั่ง else ให้ใช้ [h, p] = ttest2(A, B, 0.05, 'both', 'unqual') เพื่อรันตัวอย่าง t สองตัวอย่างในกรณีที่ความแปรปรวนไม่เท่ากัน จบคำสั่ง if/else นี้จะคำนวณ p.
สร้างคำสั่ง if/else ที่จะแสดงข้อสรุปตามค่าของ p เงื่อนไขสำหรับคำสั่ง if จะเป็น p > 0.05 เนื่องจากโดยปกติเราจะล้มเหลวในการปฏิเสธสมมติฐานว่างเมื่อค่าของ p มากกว่า 0.05 ให้ใช้คำสั่ง disp('X') เพื่อแสดง "เนื่องจากค่า p มากกว่า 0.05 เราจึงล้มเหลวในการปฏิเสธสมมติฐานว่าง " และ " ดังนั้นเราจึงล้มเหลวที่จะปฏิเสธว่าปริมาณของหลอดเลือดในสมองปกตินั้นเท่ากับของสมองที่เป็นโรคโมยาโมยะ” ในคำสั่ง else เนื่องจากโดยปกติเราจะปฏิเสธสมมติฐานว่างเมื่อค่าของ p ต่ำกว่า 0.05 ให้ใช้คำสั่ง disp('X') เพื่อแสดง "เนื่องจากค่า p น้อยกว่า 0.05 เราจึงปฏิเสธสมมติฐานว่าง " และ " ดังนั้นเราจึงล้มเหลวที่จะปฏิเสธว่าปริมาณของหลอดเลือดในสมองปกติไม่เท่ากับของสมองที่เป็นโรคโมยาโมยา” จบคำสั่ง if/else
แนะนำ:
การออกแบบเกมในการสะบัดใน 5 ขั้นตอน: 5 ขั้นตอน

การออกแบบเกมในการสะบัดใน 5 ขั้นตอน: การตวัดเป็นวิธีง่ายๆ ในการสร้างเกม โดยเฉพาะอย่างยิ่งเกมปริศนา นิยายภาพ หรือเกมผจญภัย
การตรวจจับใบหน้าบน Raspberry Pi 4B ใน 3 ขั้นตอน: 3 ขั้นตอน

การตรวจจับใบหน้าบน Raspberry Pi 4B ใน 3 ขั้นตอน: ในคำแนะนำนี้ เราจะทำการตรวจจับใบหน้าบน Raspberry Pi 4 ด้วย Shunya O/S โดยใช้ Shunyaface Library Shunyaface เป็นห้องสมุดจดจำใบหน้า/ตรวจจับใบหน้า โปรเจ็กต์นี้มีจุดมุ่งหมายเพื่อให้เกิดความเร็วในการตรวจจับและจดจำได้เร็วที่สุดด้วย
วิธีการติดตั้งปลั๊กอินใน WordPress ใน 3 ขั้นตอน: 3 ขั้นตอน
วิธีการติดตั้งปลั๊กอินใน WordPress ใน 3 ขั้นตอน: ในบทช่วยสอนนี้ ฉันจะแสดงขั้นตอนสำคัญในการติดตั้งปลั๊กอิน WordPress ให้กับเว็บไซต์ของคุณ โดยทั่วไป คุณสามารถติดตั้งปลั๊กอินได้สองวิธี วิธีแรกคือผ่าน ftp หรือผ่าน cpanel แต่ฉันจะไม่แสดงมันเพราะมันสอดคล้องกับ
การลอยแบบอะคูสติกด้วย Arduino Uno ทีละขั้นตอน (8 ขั้นตอน): 8 ขั้นตอน
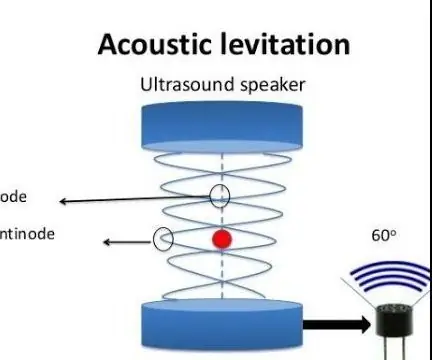
การลอยแบบอะคูสติกด้วย Arduino Uno ทีละขั้นตอน (8 ขั้นตอน): ตัวแปลงสัญญาณเสียงล้ำเสียง L298N Dc ตัวเมียอะแดปเตอร์จ่ายไฟพร้อมขา DC ตัวผู้ Arduino UNOBreadboardวิธีการทำงาน: ก่อนอื่น คุณอัปโหลดรหัสไปยัง Arduino Uno (เป็นไมโครคอนโทรลเลอร์ที่ติดตั้งดิจิตอล และพอร์ตแอนะล็อกเพื่อแปลงรหัส (C++)
เครื่อง Rube Goldberg 11 ขั้นตอน: 8 ขั้นตอน

เครื่อง 11 Step Rube Goldberg: โครงการนี้เป็นเครื่อง 11 Step Rube Goldberg ซึ่งออกแบบมาเพื่อสร้างงานง่ายๆ ในรูปแบบที่ซับซ้อน งานของโครงการนี้คือการจับสบู่ก้อนหนึ่ง
